Spatial transcriptome (Stereo-seq)
We demonstrate a noise reduction with RECODE for spatial transcriptome data. We use spatial transcriptome data of mouse embryos at E16.5, which is generated by Stereo-seq. The dataset is available from MOSTA.
We use scanpy to read/write data. Import numpy and scanpy in addlition to screcode.
[1]:
import scanpy as sc
import numpy as np
import screcode
import warnings
warnings.simplefilter('ignore')
Intel(R) Extension for Scikit-learn* enabled (https://github.com/intel/scikit-learn-intelex)
Read in the count matrix into an AnnData object.
[2]:
input_filename = 'data/E16.5_E1S1.MOSTA.h5ad'
Raw_key = "count"
adata = sc.read_h5ad("%s" % (input_filename))
adata.var_names_make_unique()
adata = adata[:,np.sum(adata.X,axis=0)>0]
adata.layers["Raw"] = adata.layers[Raw_key].toarray()
adata.layers["Proceed"] = adata.X
adata.X = adata.layers["Raw"]
adata
[2]:
AnnData object with n_obs × n_vars = 121767 × 28204
obs: 'n_genes_by_counts', 'log1p_n_genes_by_counts', 'total_counts', 'log1p_total_counts', 'annotation', 'Regulon - AI987944', 'Regulon - Alx1', 'Regulon - Alx4', 'Regulon - Arid3a', 'Regulon - Arnt2', 'Regulon - Arx', 'Regulon - Atf1', 'Regulon - Atf2', 'Regulon - Atf3', 'Regulon - Atf4', 'Regulon - Atf6', 'Regulon - Atoh1', 'Regulon - Bach2', 'Regulon - Barhl1', 'Regulon - Barhl2', 'Regulon - Barx1', 'Regulon - Bcl6', 'Regulon - Bclaf1', 'Regulon - Bdp1', 'Regulon - Bhlhe40', 'Regulon - Bmyc', 'Regulon - Brca1', 'Regulon - Brf1', 'Regulon - Brf2', 'Regulon - Cdx1', 'Regulon - Cdx2', 'Regulon - Cebpa', 'Regulon - Cebpb', 'Regulon - Cebpe', 'Regulon - Cic', 'Regulon - Clock', 'Regulon - Creb1', 'Regulon - Creb3l1', 'Regulon - Creb3l2', 'Regulon - Creb5', 'Regulon - Crebl2', 'Regulon - Ctcf', 'Regulon - Ctcfl', 'Regulon - Cux1', 'Regulon - Dbp', 'Regulon - Dbx1', 'Regulon - Ddit3', 'Regulon - Dlx1', 'Regulon - Dlx2', 'Regulon - Dlx3', 'Regulon - Dlx5', 'Regulon - Dlx6', 'Regulon - Dmbx1', 'Regulon - E2f1', 'Regulon - E2f2', 'Regulon - E2f3', 'Regulon - E2f4', 'Regulon - E2f5', 'Regulon - E2f6', 'Regulon - E2f7', 'Regulon - E2f8', 'Regulon - Ebf1', 'Regulon - Egr1', 'Regulon - Egr3', 'Regulon - Elf1', 'Regulon - Elf2', 'Regulon - Elf3', 'Regulon - Elf4', 'Regulon - Elk3', 'Regulon - Elk4', 'Regulon - Emx1', 'Regulon - Emx2', 'Regulon - En1', 'Regulon - En2', 'Regulon - Ep300', 'Regulon - Erg', 'Regulon - Esrra', 'Regulon - Esrrg', 'Regulon - Ets1', 'Regulon - Ets2', 'Regulon - Etv6', 'Regulon - Evx1', 'Regulon - Ezh2', 'Regulon - Fli1', 'Regulon - Fos', 'Regulon - Fosl1', 'Regulon - Fosl2', 'Regulon - Foxa1', 'Regulon - Foxa2', 'Regulon - Foxa3', 'Regulon - Foxc1', 'Regulon - Foxc2', 'Regulon - Foxd1', 'Regulon - Foxd2', 'Regulon - Foxd3', 'Regulon - Foxf1', 'Regulon - Foxf2', 'Regulon - Foxg1', 'Regulon - Foxi1', 'Regulon - Foxj1', 'Regulon - Foxj2', 'Regulon - Foxk1', 'Regulon - Foxl1', 'Regulon - Foxl2', 'Regulon - Foxn2', 'Regulon - Foxn3', 'Regulon - Foxo1', 'Regulon - Foxo3', 'Regulon - Foxo4', 'Regulon - Foxp1', 'Regulon - Foxp2', 'Regulon - Foxp3', 'Regulon - Foxp4', 'Regulon - Foxq1', 'Regulon - Gabpa', 'Regulon - Gabpb1', 'Regulon - Gata1', 'Regulon - Gata2', 'Regulon - Gata3', 'Regulon - Gata4', 'Regulon - Gata5', 'Regulon - Gata6', 'Regulon - Gbx1', 'Regulon - Gbx2', 'Regulon - Gli1', 'Regulon - Gli2', 'Regulon - Glis2', 'Regulon - Gm12845', 'Regulon - Gm14327', 'Regulon - Gm28308', 'Regulon - Gm38394', 'Regulon - Gmeb2', 'Regulon - Grhl1', 'Regulon - Grhl2', 'Regulon - Grhl3', 'Regulon - Gsx1', 'Regulon - Gsx2', 'Regulon - Gtf2f1', 'Regulon - Gtf3c2', 'Regulon - Hcfc1', 'Regulon - Hdac2', 'Regulon - Hes1', 'Regulon - Hes5', 'Regulon - Hif1a', 'Regulon - Hinfp', 'Regulon - Hmga2', 'Regulon - Hmgb3', 'Regulon - Hmgn3', 'Regulon - Hmx1', 'Regulon - Hmx3', 'Regulon - Hnf1b', 'Regulon - Hnf4a', 'Regulon - Hnf4g', 'Regulon - Hoxa10', 'Regulon - Hoxa11', 'Regulon - Hoxa2', 'Regulon - Hoxa3', 'Regulon - Hoxa4', 'Regulon - Hoxa5', 'Regulon - Hoxa7', 'Regulon - Hoxa9', 'Regulon - Hoxb13', 'Regulon - Hoxb3', 'Regulon - Hoxb4', 'Regulon - Hoxb5', 'Regulon - Hoxb6', 'Regulon - Hoxb7', 'Regulon - Hoxb8', 'Regulon - Hoxb9', 'Regulon - Hoxc10', 'Regulon - Hoxc11', 'Regulon - Hoxc13', 'Regulon - Hoxc4', 'Regulon - Hoxc5', 'Regulon - Hoxc6', 'Regulon - Hoxc8', 'Regulon - Hoxc9', 'Regulon - Hoxd1', 'Regulon - Hoxd10', 'Regulon - Hoxd11', 'Regulon - Hoxd12', 'Regulon - Hoxd13', 'Regulon - Hoxd3', 'Regulon - Hoxd8', 'Regulon - Hoxd9', 'Regulon - Hsf2', 'Regulon - Ikzf1', 'Regulon - Irf2', 'Regulon - Irf5', 'Regulon - Irf6', 'Regulon - Irf8', 'Regulon - Irx3', 'Regulon - Isl1', 'Regulon - Isl2', 'Regulon - Jdp2', 'Regulon - Jun', 'Regulon - Junb', 'Regulon - Jund', 'Regulon - Kdm4a', 'Regulon - Kdm5a', 'Regulon - Kdm5b', 'Regulon - Klf10', 'Regulon - Klf13', 'Regulon - Klf15', 'Regulon - Klf16', 'Regulon - Klf2', 'Regulon - Klf3', 'Regulon - Klf4', 'Regulon - Klf5', 'Regulon - Klf6', 'Regulon - Klf7', 'Regulon - Klf8', 'Regulon - Klf9', 'Regulon - Lbx1', 'Regulon - Lef1', 'Regulon - Lhx1', 'Regulon - Lhx2', 'Regulon - Lhx5', 'Regulon - Lhx6', 'Regulon - Lhx8', 'Regulon - Lhx9', 'Regulon - Lmo2', 'Regulon - Lmx1a', 'Regulon - Lmx1b', 'Regulon - Ltf', 'Regulon - Maf', 'Regulon - Mafa', 'Regulon - Mafb', 'Regulon - Mafk', 'Regulon - Max', 'Regulon - Maz', 'Regulon - Mbd1', 'Regulon - Mef2a', 'Regulon - Mef2c', 'Regulon - Mef2d', 'Regulon - Meis1', 'Regulon - Meis2', 'Regulon - Meis3', 'Regulon - Mnx1', 'Regulon - Msc', 'Regulon - Msx1', 'Regulon - Msx2', 'Regulon - Msx3', 'Regulon - Mxi1', 'Regulon - Myb', 'Regulon - Mybl1', 'Regulon - Mybl2', 'Regulon - Myc', 'Regulon - Myf5', 'Regulon - Myf6', 'Regulon - Mynn', 'Regulon - Myod1', 'Regulon - Myog', 'Regulon - Nanos1', 'Regulon - Nelfe', 'Regulon - Neurod1', 'Regulon - Nfatc1', 'Regulon - Nfatc2', 'Regulon - Nfatc3', 'Regulon - Nfatc4', 'Regulon - Nfe2', 'Regulon - Nfe2l1', 'Regulon - Nfe2l2', 'Regulon - Nfic', 'Regulon - Nfil3', 'Regulon - Nfkb1', 'Regulon - Nfkb2', 'Regulon - Nfyb', 'Regulon - Nfyc', 'Regulon - Nhlh2', 'Regulon - Nkx1-1', 'Regulon - Nkx2-1', 'Regulon - Nkx2-4', 'Regulon - Nkx2-5', 'Regulon - Nkx6-1', 'Regulon - Nkx6-2', 'Regulon - Nkx6-3', 'Regulon - Nobox', 'Regulon - Npdc1', 'Regulon - Nr1h3', 'Regulon - Nr1h4', 'Regulon - Nr1i2', 'Regulon - Nr2c1', 'Regulon - Nr2c2', 'Regulon - Nr2f2', 'Regulon - Nr3c1', 'Regulon - Nr4a1', 'Regulon - Nr5a1', 'Regulon - Nr5a2', 'Regulon - Nrf1', 'Regulon - Nucb1', 'Regulon - Olig1', 'Regulon - Olig3', 'Regulon - Onecut1', 'Regulon - Onecut2', 'Regulon - Onecut3', 'Regulon - Otx2', 'Regulon - Ovol2', 'Regulon - Patz1', 'Regulon - Pax5', 'Regulon - Pax6', 'Regulon - Pbx2', 'Regulon - Pbx3', 'Regulon - Pdx1', 'Regulon - Phf8', 'Regulon - Phox2a', 'Regulon - Phox2b', 'Regulon - Pitx1', 'Regulon - Pitx2', 'Regulon - Pknox1', 'Regulon - Plagl1', 'Regulon - Pml', 'Regulon - Pole3', 'Regulon - Pou2f1', 'Regulon - Pou3f1', 'Regulon - Pou3f2', 'Regulon - Pou3f3', 'Regulon - Pou3f4', 'Regulon - Pou4f1', 'Regulon - Pou4f2', 'Regulon - Pou4f3', 'Regulon - Pou6f1', 'Regulon - Ppara', 'Regulon - Pparg', 'Regulon - Ppargc1a', 'Regulon - Prdm16', 'Regulon - Prop1', 'Regulon - Prrx2', 'Regulon - Prrxl1', 'Regulon - Psmd12', 'Regulon - Rad21', 'Regulon - Rara', 'Regulon - Rarb', 'Regulon - Rarg', 'Regulon - Rax', 'Regulon - Rbbp5', 'Regulon - Rcor1', 'Regulon - Rela', 'Regulon - Relb', 'Regulon - Rest', 'Regulon - Rfx2', 'Regulon - Rfx3', 'Regulon - Rfx4', 'Regulon - Rfxap', 'Regulon - Rreb1', 'Regulon - Runx1', 'Regulon - Runx3', 'Regulon - Rxra', 'Regulon - Rxrb', 'Regulon - Rxrg', 'Regulon - Sap30', 'Regulon - Scx', 'Regulon - Setdb1', 'Regulon - Shox2', 'Regulon - Sin3a', 'Regulon - Six1', 'Regulon - Six2', 'Regulon - Six4', 'Regulon - Smad1', 'Regulon - Smad5', 'Regulon - Smarca4', 'Regulon - Smarcb1', 'Regulon - Smarcc2', 'Regulon - Smc3', 'Regulon - Snai3', 'Regulon - Sox10', 'Regulon - Sox11', 'Regulon - Sox12', 'Regulon - Sox13', 'Regulon - Sox15', 'Regulon - Sox17', 'Regulon - Sox18', 'Regulon - Sox2', 'Regulon - Sox21', 'Regulon - Sox3', 'Regulon - Sox4', 'Regulon - Sox7', 'Regulon - Sox8', 'Regulon - Sox9', 'Regulon - Sp1', 'Regulon - Sp2', 'Regulon - Sp3', 'Regulon - Sp4', 'Regulon - Sp6', 'Regulon - Sp7', 'Regulon - Sp8', 'Regulon - Sp9', 'Regulon - Spdef', 'Regulon - Spi1', 'Regulon - Srebf1', 'Regulon - Srebf2', 'Regulon - Srf', 'Regulon - Stat1', 'Regulon - Stat3', 'Regulon - Stat6', 'Regulon - Supt20', 'Regulon - Taf1', 'Regulon - Tagln2', 'Regulon - Tal1', 'Regulon - Tbp', 'Regulon - Tbx1', 'Regulon - Tbx15', 'Regulon - Tbx2', 'Regulon - Tbx4', 'Regulon - Tbx5', 'Regulon - Tcf7', 'Regulon - Tcf7l1', 'Regulon - Tcf7l2', 'Regulon - Tead1', 'Regulon - Tead2', 'Regulon - Tead3', 'Regulon - Tead4', 'Regulon - Tef', 'Regulon - Tfap2a', 'Regulon - Tfcp2l1', 'Regulon - Tfdp1', 'Regulon - Tff3', 'Regulon - Thap1', 'Regulon - Thra', 'Regulon - Thrb', 'Regulon - Tlx2', 'Regulon - Tlx3', 'Regulon - Traf4', 'Regulon - Trim28', 'Regulon - Trp53', 'Regulon - Trp63', 'Regulon - Trp73', 'Regulon - Twist1', 'Regulon - Twist2', 'Regulon - Ubtf', 'Regulon - Uncx', 'Regulon - Usf2', 'Regulon - Vax1', 'Regulon - Vezf1', 'Regulon - Vsx2', 'Regulon - Wt1', 'Regulon - Xbp1', 'Regulon - Xrcc4', 'Regulon - Yy1', 'Regulon - Zbtb14', 'Regulon - Zbtb7b', 'Regulon - Zeb1', 'Regulon - Zfhx2', 'Regulon - Zfhx3', 'Regulon - Zfp110', 'Regulon - Zfp112', 'Regulon - Zfp14', 'Regulon - Zfp143', 'Regulon - Zfp146', 'Regulon - Zfp148', 'Regulon - Zfp189', 'Regulon - Zfp266', 'Regulon - Zfp281', 'Regulon - Zfp369', 'Regulon - Zfp467', 'Regulon - Zfp612', 'Regulon - Zfp641', 'Regulon - Zfp647', 'Regulon - Zfp652', 'Regulon - Zfp672', 'Regulon - Zfp708', 'Regulon - Zfp710', 'Regulon - Zfp729b', 'Regulon - Zfp740', 'Regulon - Zfp950', 'Regulon - Zfp974', 'Regulon - Zfp975', 'Regulon - Zfx', 'Regulon - Zic1', 'Regulon - Zic2', 'Regulon - Zic3', 'Regulon - Zic4', 'Regulon - Zmat4', 'Regulon - Zmiz1', 'Module_1', 'Module_2', 'Module_3', 'Module_4', 'Module_5', 'Module_6', 'Module_7', 'Module_8', 'Module_9', 'Module_10', 'Module_11', 'Module_12', 'Module_13', 'Module_14', 'Module_15', 'Module_16', 'Module_17', 'Module_18', 'Module_19', 'Module_20', 'Module_21', 'Module_22', 'Module_23', 'Module_24', 'Module_25', 'Module_26', 'Module_27', 'Module_28', 'Module_29', 'Module_30', 'Module_31', 'Module_32', 'Module_33', 'Module_34', 'Module_35', 'pct_counts_in_top_50_genes', 'pct_counts_in_top_100_genes', 'pct_counts_in_top_200_genes', 'pct_counts_in_top_500_genes'
var: 'n_cells', 'n_cells_by_counts', 'mean_counts', 'log1p_mean_counts', 'pct_dropout_by_counts', 'total_counts', 'log1p_total_counts', 'Regulon - Alx4', 'Regulon - Arnt2', 'Regulon - Atf1', 'Regulon - Atf3', 'Regulon - Atf4', 'Regulon - Bach2', 'Regulon - Barhl1', 'Regulon - Barhl2', 'Regulon - Barx1', 'Regulon - Bclaf1', 'Regulon - Brca1', 'Regulon - Cdx1', 'Regulon - Cdx2', 'Regulon - Cebpa', 'Regulon - Cebpb', 'Regulon - Cebpe', 'Regulon - Clock', 'Regulon - Creb1', 'Regulon - Creb3l2', 'Regulon - Ctcfl', 'Regulon - Cux1', 'Regulon - Dbp', 'Regulon - Ddit3', 'Regulon - Dlx1', 'Regulon - Dlx2', 'Regulon - Dlx3', 'Regulon - Dlx5', 'Regulon - Dlx6', 'Regulon - Dmbx1', 'Regulon - E2f1', 'Regulon - E2f3', 'Regulon - E2f4', 'Regulon - E2f5', 'Regulon - E2f6', 'Regulon - E2f7', 'Regulon - E2f8', 'Regulon - Egr1', 'Regulon - Egr3', 'Regulon - Elf2', 'Regulon - Elf4', 'Regulon - Elk3', 'Regulon - Elk4', 'Regulon - Emx1', 'Regulon - Emx2', 'Regulon - En2', 'Regulon - Ep300', 'Regulon - Esrra', 'Regulon - Esrrg', 'Regulon - Ets1', 'Regulon - Ets2', 'Regulon - Etv6', 'Regulon - Ezh2', 'Regulon - Fli1', 'Regulon - Fos', 'Regulon - Fosl2', 'Regulon - Foxa1', 'Regulon - Foxa2', 'Regulon - Foxa3', 'Regulon - Foxc1', 'Regulon - Foxc2', 'Regulon - Foxf1', 'Regulon - Foxf2', 'Regulon - Foxi1', 'Regulon - Foxj1', 'Regulon - Foxj2', 'Regulon - Foxl1', 'Regulon - Foxl2', 'Regulon - Foxn2', 'Regulon - Foxo1', 'Regulon - Foxo3', 'Regulon - Foxo4', 'Regulon - Foxp1', 'Regulon - Foxp2', 'Regulon - Foxp4', 'Regulon - Gabpa', 'Regulon - Gata1', 'Regulon - Gata2', 'Regulon - Gata3', 'Regulon - Gata4', 'Regulon - Gata5', 'Regulon - Gata6', 'Regulon - Gbx2', 'Regulon - Gli1', 'Regulon - Gli2', 'Regulon - Glis2', 'Regulon - Gm38394', 'Regulon - Gmeb2', 'Regulon - Grhl1', 'Regulon - Grhl2', 'Regulon - Grhl3', 'Regulon - Gsx1', 'Regulon - Hdac2', 'Regulon - Hes1', 'Regulon - Hinfp', 'Regulon - Hmga2', 'Regulon - Hmgb3', 'Regulon - Hnf1b', 'Regulon - Hnf4a', 'Regulon - Hnf4g', 'Regulon - Hoxa10', 'Regulon - Hoxa11', 'Regulon - Hoxa2', 'Regulon - Hoxa3', 'Regulon - Hoxa5', 'Regulon - Hoxa7', 'Regulon - Hoxa9', 'Regulon - Hoxb3', 'Regulon - Hoxb4', 'Regulon - Hoxb5', 'Regulon - Hoxb6', 'Regulon - Hoxb7', 'Regulon - Hoxb8', 'Regulon - Hoxb9', 'Regulon - Hoxc10', 'Regulon - Hoxc11', 'Regulon - Hoxc13', 'Regulon - Hoxc6', 'Regulon - Hoxc9', 'Regulon - Hoxd11', 'Regulon - Hoxd12', 'Regulon - Hoxd13', 'Regulon - Hoxd9', 'Regulon - Hsf2', 'Regulon - Ikzf1', 'Regulon - Irf2', 'Regulon - Irf5', 'Regulon - Irf6', 'Regulon - Irf8', 'Regulon - Isl1', 'Regulon - Isl2', 'Regulon - Jun', 'Regulon - Junb', 'Regulon - Jund', 'Regulon - Klf2', 'Regulon - Klf3', 'Regulon - Klf4', 'Regulon - Klf5', 'Regulon - Klf6', 'Regulon - Klf8', 'Regulon - Lef1', 'Regulon - Lhx1', 'Regulon - Lhx2', 'Regulon - Lhx5', 'Regulon - Lhx6', 'Regulon - Lhx8', 'Regulon - Lhx9', 'Regulon - Lmx1a', 'Regulon - Ltf', 'Regulon - Maf', 'Regulon - Mafb', 'Regulon - Mafk', 'Regulon - Mbd1', 'Regulon - Mef2a', 'Regulon - Mef2c', 'Regulon - Mef2d', 'Regulon - Mnx1', 'Regulon - Msc', 'Regulon - Mxi1', 'Regulon - Myb', 'Regulon - Mybl1', 'Regulon - Mybl2', 'Regulon - Myc', 'Regulon - Myf5', 'Regulon - Myf6', 'Regulon - Mynn', 'Regulon - Myod1', 'Regulon - Myog', 'Regulon - Nanos1', 'Regulon - Neurod1', 'Regulon - Nfatc1', 'Regulon - Nfatc4', 'Regulon - Nfe2', 'Regulon - Nfic', 'Regulon - Nfkb2', 'Regulon - Nfyb', 'Regulon - Nkx1-1', 'Regulon - Nkx6-2', 'Regulon - Nr1h3', 'Regulon - Nr1i2', 'Regulon - Nr2c1', 'Regulon - Nr4a1', 'Regulon - Nr5a1', 'Regulon - Nr5a2', 'Regulon - Nucb1', 'Regulon - Onecut1', 'Regulon - Onecut2', 'Regulon - Onecut3', 'Regulon - Otx2', 'Regulon - Ovol2', 'Regulon - Pax6', 'Regulon - Pdx1', 'Regulon - Phox2a', 'Regulon - Phox2b', 'Regulon - Pitx2', 'Regulon - Pou2f1', 'Regulon - Pou3f1', 'Regulon - Pou3f3', 'Regulon - Pou4f3', 'Regulon - Pparg', 'Regulon - Prrx2', 'Regulon - Rara', 'Regulon - Rarb', 'Regulon - Rax', 'Regulon - Rela', 'Regulon - Rfx2', 'Regulon - Rfx3', 'Regulon - Rfx4', 'Regulon - Runx1', 'Regulon - Rxra', 'Regulon - Shox2', 'Regulon - Six1', 'Regulon - Six2', 'Regulon - Six4', 'Regulon - Smad1', 'Regulon - Smad5', 'Regulon - Smarcc2', 'Regulon - Snai3', 'Regulon - Sox10', 'Regulon - Sox11', 'Regulon - Sox12', 'Regulon - Sox13', 'Regulon - Sox17', 'Regulon - Sox18', 'Regulon - Sox2', 'Regulon - Sox21', 'Regulon - Sox4', 'Regulon - Sox7', 'Regulon - Sox9', 'Regulon - Sp1', 'Regulon - Sp3', 'Regulon - Sp4', 'Regulon - Sp7', 'Regulon - Spdef', 'Regulon - Spi1', 'Regulon - Srebf2', 'Regulon - Srf', 'Regulon - Stat6', 'Regulon - Supt20', 'Regulon - Taf1', 'Regulon - Tagln2', 'Regulon - Tal1', 'Regulon - Tbp', 'Regulon - Tbx2', 'Regulon - Tbx5', 'Regulon - Tcf7', 'Regulon - Tcf7l1', 'Regulon - Tcf7l2', 'Regulon - Tead1', 'Regulon - Tead2', 'Regulon - Tead3', 'Regulon - Tead4', 'Regulon - Tfap2a', 'Regulon - Tfcp2l1', 'Regulon - Tfdp1', 'Regulon - Tff3', 'Regulon - Thap1', 'Regulon - Tlx2', 'Regulon - Tlx3', 'Regulon - Trp53', 'Regulon - Trp63', 'Regulon - Twist1', 'Regulon - Uncx', 'Regulon - Xbp1', 'Regulon - Yy1', 'Regulon - Zbtb14', 'Regulon - Zfp112', 'Regulon - Zfp14', 'Regulon - Zfp189', 'Regulon - Zfp641', 'Regulon - Zfp672', 'Regulon - Zfp729b', 'Regulon - Zfp740', 'Regulon - Zfp974', 'Regulon - Zfp975', 'Regulon - Zic2', 'Regulon - Zmat4', 'Module_1', 'Module_10', 'Module_11', 'Module_12', 'Module_13', 'Module_14', 'Module_15', 'Module_16', 'Module_17', 'Module_18', 'Module_19', 'Module_2', 'Module_20', 'Module_21', 'Module_22', 'Module_23', 'Module_24', 'Module_25', 'Module_26', 'Module_27', 'Module_28', 'Module_29', 'Module_3', 'Module_30', 'Module_31', 'Module_32', 'Module_33', 'Module_34', 'Module_35', 'Module_4', 'Module_5', 'Module_6', 'Module_7', 'Module_8', 'Module_9'
uns: 'annotation_colors'
obsm: 'spatial'
varm: 'PCs'
layers: 'count', 'Raw', 'Proceed'
[3]:
import matplotlib.pyplot as plt
import matplotlib.cm as cm
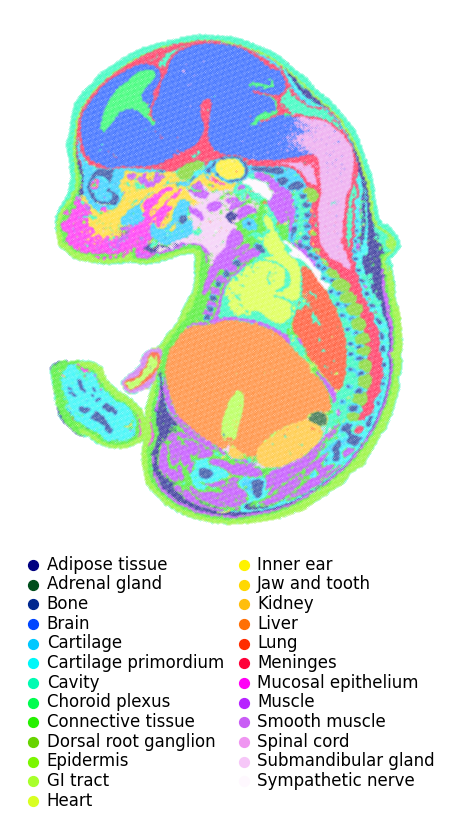
clusters = adata.obs["annotation"]
cluster_set = np.unique(clusters)
sp_x, sp_y = adata.obsm["spatial"][:,0], -adata.obsm["spatial"][:,1]
psize = 0.02
figsize=(5,7)
colors = cm.get_cmap('gist_ncar', 25)
fig,ax = plt.subplots(figsize=figsize)
for i in range(len(cluster_set)):
idx_ = clusters == cluster_set[i]
ax.scatter(sp_x[idx_], sp_y[idx_],s=psize,label=cluster_set[i], color=colors(i))
ax.axis('off')
ax.legend(markerscale=50,bbox_to_anchor=(0.5, 0), loc='upper center', borderaxespad=0, fontsize=12,ncol=2,columnspacing=0.2,labelspacing=0.1,handletextpad=-0.2,frameon=False)
[3]:
<matplotlib.legend.Legend at 0x178cf3e8880>
[4]:
import matplotlib.cm as cm
import math
clusters = adata.obs["annotation"]
cluster_set = np.unique(clusters)
sp_x, sp_y = adata.obsm["spatial"][:,0], -adata.obsm["spatial"][:,1]
psize = 0.5
figsize=(2.5,3.5)
n_cols = 10
n_rows = math.ceil(len(cluster_set)/n_cols)
colors = cm.get_cmap('gist_ncar', 25)
fig,ax = plt.subplots(n_rows,n_cols,figsize=(figsize[0]*n_cols,figsize[1]*n_rows))
[[ax[i,j].axis('off') for i in range(n_rows)] for j in range(n_cols)]
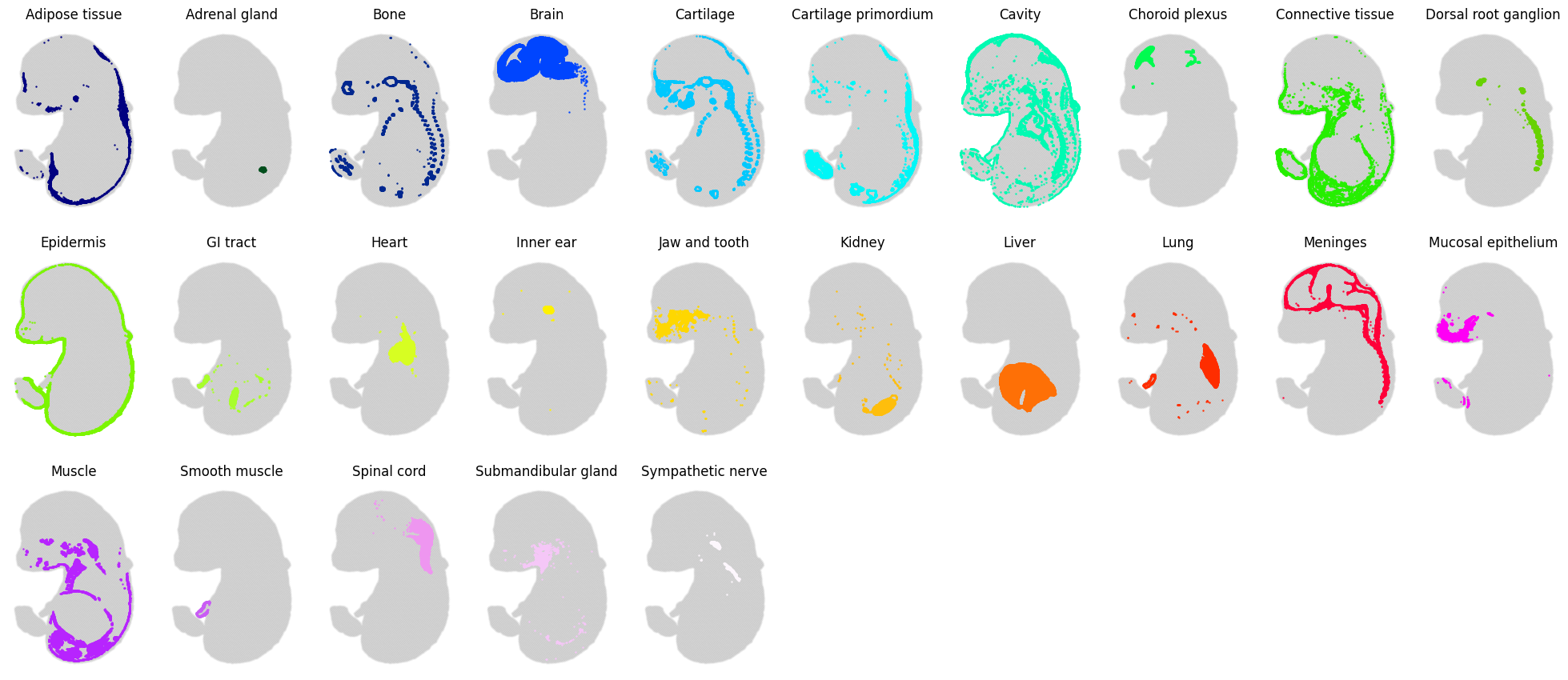
for i in range(len(cluster_set)):
idx_ = clusters == cluster_set[i]
row_ = int(i / n_cols)
col_ = i % n_cols
ax_ = ax[row_,col_]
ax_.scatter(sp_x, sp_y,s=0.01,color="gray",alpha=0.1)
ax_.scatter(sp_x[idx_], sp_y[idx_],s=psize,color=colors(i))
ax_.axis('off')
ax_.set_title(cluster_set[i])
Apply RECODE
Apply RECODE to the count matrix (without using spatial coordinates).
[5]:
recode = screcode.RECODE(seq_target='RNA',version=2,fast_algorithm_ell_ub=3500)
adata = recode.fit_transform(adata)
start RECODE for scRNA-seq data
end RECODE for scRNA-seq
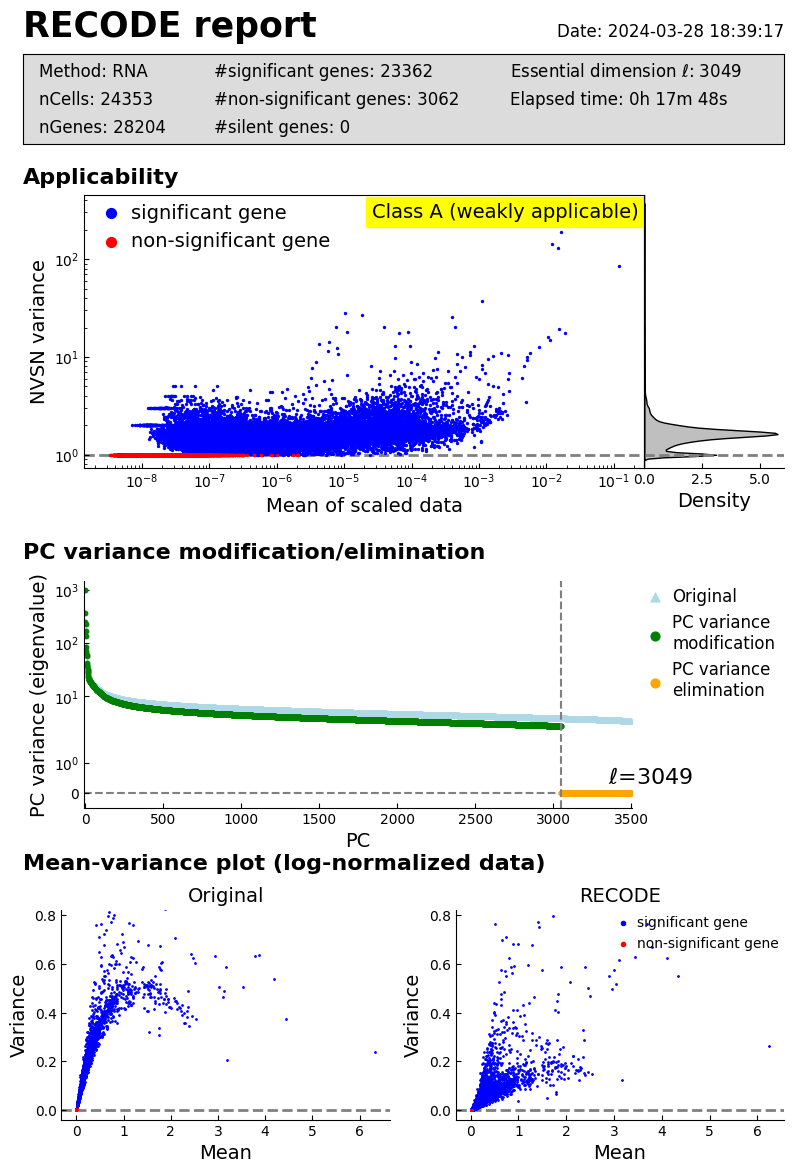
log: {'seq_target': 'RNA', '#significant genes': 23362, '#non-significant genes': 3062, '#silent genes': 0, 'ell': 3049, 'Elapsed time': '0h 17m 48s 400ms', 'solver': 'randomized', '#test_data': 24353}
Performance check
[6]:
recode.report()
Log normalizaation
[7]:
target_sum = 1e+4
adata = recode.lognormalize(adata,target_sum=target_sum)
Normalized data are stored in "RECODE_norm" and "RECODE_log"
[8]:
adata.layers["Raw_norm"] = target_sum*adata.layers[Raw_key].toarray()/np.sum(adata.layers[Raw_key].toarray(),axis=1)[:,np.newaxis]
adata.layers["Raw_log"] = np.log(adata.layers["Raw_norm"]+1)
Plot spatial gene expression
[10]:
def spatial_gex(
genes,
sp_x = adata.obsm["spatial"][:,0],
sp_y = -adata.obsm["spatial"][:,1],
psize = 1,
figsize=(2.5,3.5),
dpi=100,
percentiles = [0,99],
fs_title = 20,
fs_label = 20,
):
fig,ax = plt.subplots(2,len(genes),figsize=(figsize[0]*len(genes),figsize[1]*2),tight_layout=True)
for i in range(len(genes)):
idx_gene = adata.var.index == genes[i]
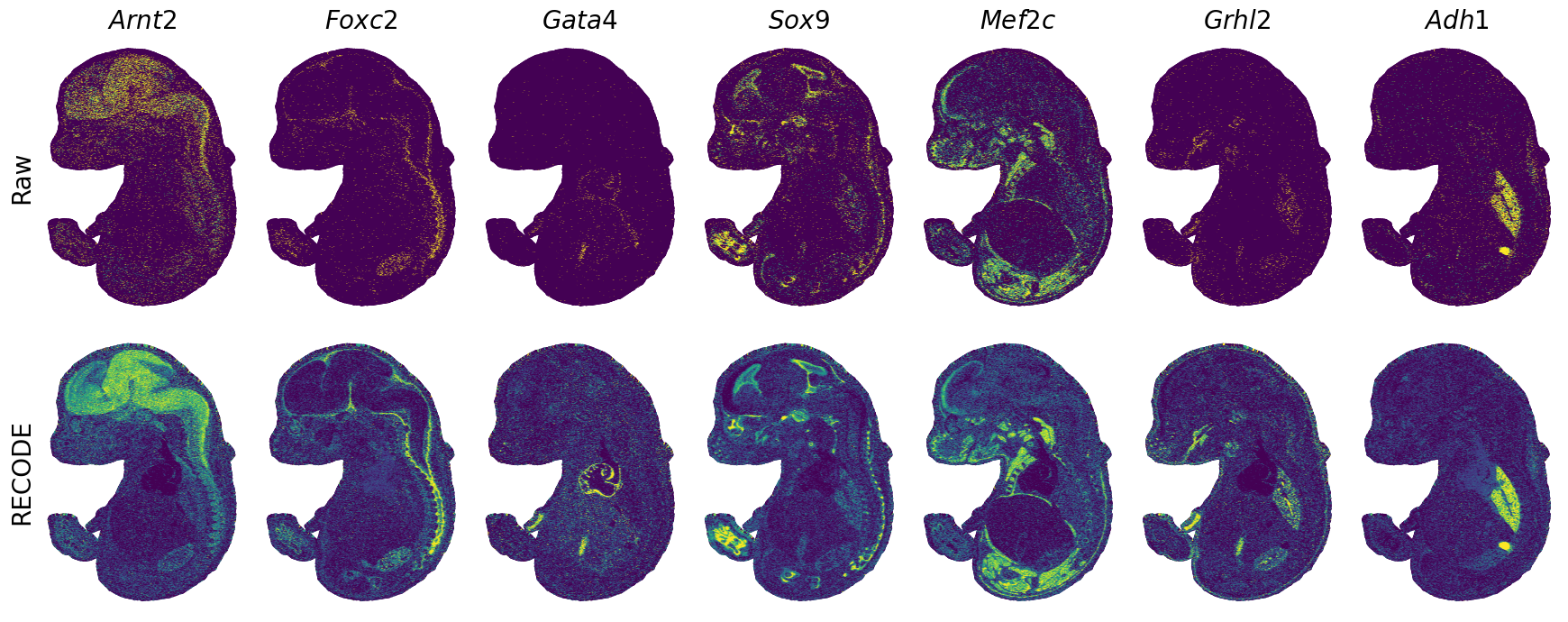
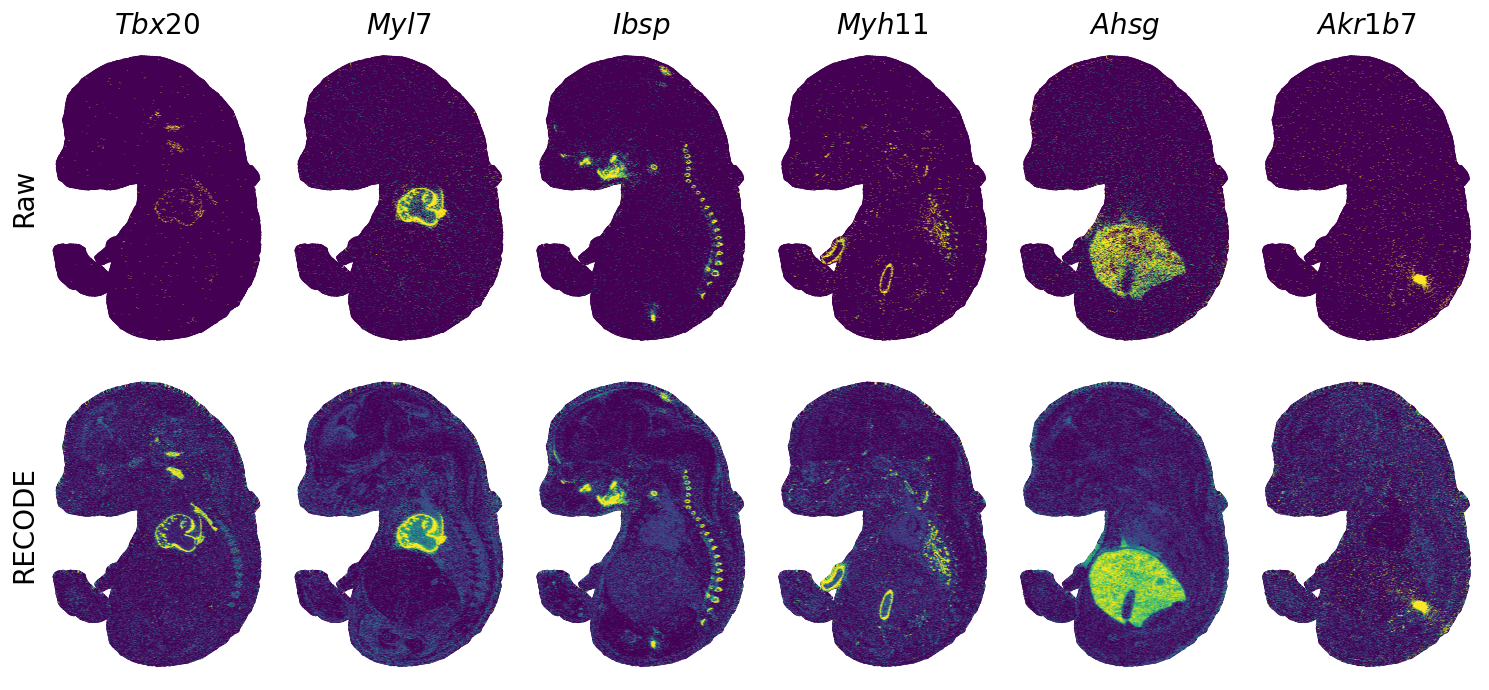
exp = adata.layers["RECODE_log"][:,idx_gene]
vmin,vmax = np.percentile(exp,percentiles[0]),np.percentile(exp,percentiles[1])
ax_ = ax[1,i]
ax_.scatter(sp_x, sp_y,c=exp,s=psize,marker="H",vmin=vmin,vmax=vmax)
if i== 0:
ax_.set_ylabel("RECODE",fontsize=fs_label)
ax_.tick_params(bottom=False, left=False, right=False, top=False,
labelbottom=False, labelleft=False, labelright=False, labeltop=False)
[ax_.spines[c_].set_visible(False) for c_ in ['right','top','bottom','left']]
else:
ax_.axis('off')
exp = adata.layers["Raw_log"][:,idx_gene]
ax_ = ax[0,i]
ax_.scatter(sp_x, sp_y,c=exp,s=psize,marker="H",vmin=vmin,vmax=vmax)
ax_.set_title("$\it{%s}$" % genes[i],fontsize=fs_title)
if i== 0:
ax_.set_ylabel("Raw",fontsize=fs_label)
ax_.tick_params(bottom=False, left=False, right=False, top=False,
labelbottom=False, labelleft=False, labelright=False, labeltop=False)
[ax_.spines[c_].set_visible(False) for c_ in ['right','top','bottom','left']]
else:
ax_.axis('off')
spatial_gex(genes = ["Arnt2", "Foxc2", "Gata4", "Sox9", "Mef2c", "Grhl2", "Adh1"])
spatial_gex(genes = ["Tbx20","Myl7","Ibsp", "Myh11", "Ahsg", "Akr1b7"])